ここではGitHubからCOVID-19(新型コロナ)の生データをダウンロードしてPython, Pandas, Matplotlibで解析する方法を説明します。
当サイトではPythonのコードを記述するときMicrosoftの「Visual Studio Code(VSC)」を使用しています。
今回からVSCの「Python Interactive window」
を使用してJupter(IPython Notebook)のような環境で説明します。
ちなみに、VSCを通常の環境からインタラクティブな環境に切り換えるにはコードを記述するときコメント「# %%」を入力します。
詳しい、操作手順については「ここ」
を参照してください。
インタラクティブな環境では、Pythonの「print(), plt.show()」などを使う必要がないので掲載しているコードでは省略しています。
VSCで通常の環境で使用するときは、必要に応じて「print(), plt.show()」等を追加してください。
この記事では、Pandas、Matplotlibのライブラリを使用しますので
「記事(Article001) | 記事(Article002) | 記事(Article003) | 記事(Article004)」
を参照して事前にインストールしておいてください。
Pythonのコードを入力するときにMicrosoftのVisula Studio Codeを使用します。
まだ、インストールしていないときは「記事(Article001)」を参照してインストールしておいてください。
説明文の左側に図の画像が表示されていますが縮小されています。
画像を拡大するにはマウスを画像上に移動してクリックします。
画像が拡大表示されます。拡大された画像を閉じるには右上の[X]をクリックします。
画像の任意の場所をクリックして閉じることもできます。
-
Visual Studio Codeを起動してインタラクティブな環境にする
Visual Studio Code(VSC)を起動して新規ファイル(*.py)を作成したら以下のようなコメント「# %%」を入力します。
するとインタラクティブな環境に切り換わって右側に専用のウィンドウが表示されます。
右側のインタラクティブ・ウィンドウからはPythonのコードを入力して[Shift+Enter]で実行させることもできます。
左側のコードを実行させるには「Run Cell」「Run Below」「Run Above」等をクリックします。
特定のセル(「# %%」で囲まれた範囲のこと)を選択して[Ctrl+Enter]で実行させることもできます。
# Data Source: https://github.com/owid/covid-19-data/tree/master/public/data/
# csv file: data/csv/article012/owid-covid-data.csv
# %%
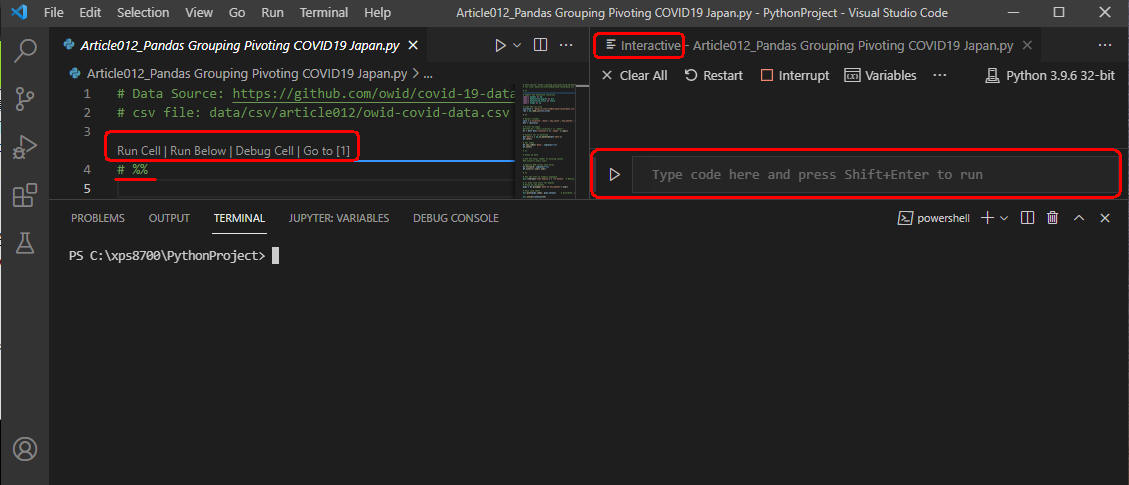 図1
図1は実行結果です。VSCの右側に「Interactive」専用のウィンドウが表示されています。
図1
図1は実行結果です。VSCの右側に「Interactive」専用のウィンドウが表示されています。
-
GutHubからダウンロードしたCOVID-19のデータを取り込む
行2-5ではPythonのライブラリを取り込んでいます。
行8ではGitHubからダウンロードしたCOVID-19のファイルのパスを定義しています。
行9ではPandasのread_csv()メソッドでCSVファイルを取り込んでDataFrameに格納しています。
左側のコードウィンドウから[Run Cell]をクリックしてセルのコードを実行します。
マウスをセル内に移動して[Ctrl+Enter]で実行させることもできます。
右側のインタラクティブウィンドウの最下位に「raw.info()」を入力して実行ボタン[▶]をクリックします。
[Shit+Enter]で実行させることもできます。
DataFrameの情報が表示されたら、レコード件数(115,872)と列数(62)を確認します。
さらに、列「date」がobject型(str型)になっていることも確認します。
# Import the necessary libraries
import pandas as pd
import matplotlib.pyplot as plt
import matplotlib.style as style
import numpy as np
# Load the csv file
csv_file = 'data/csv/article012/owid-covid-data.csv'
raw = pd.read_csv(csv_file)
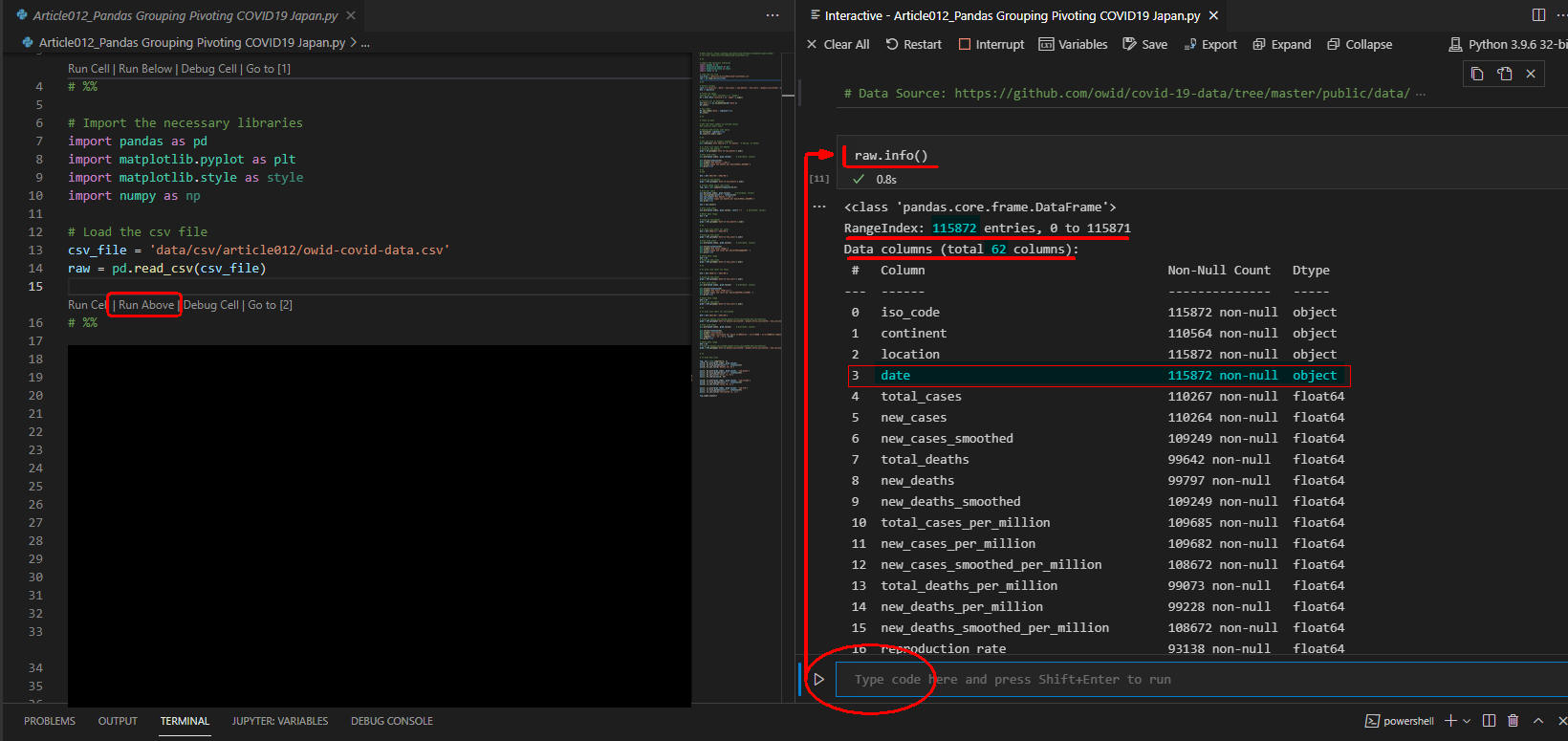 図2
図2は「raw.info()」の実行結果です。
PandasのDataFrameの情報が表示されています。
列3の「date」がdatetime型ではなくobject型になっています。
図2
図2は「raw.info()」の実行結果です。
PandasのDataFrameの情報が表示されています。
列3の「date」がdatetime型ではなくobject型になっています。
-
ダウンロードしたデータを絞り込む
「Run Cell」をクリックしてセル内のコードを実行させます。
行2-3ではDataFrameの列数を62から7に絞り込んでいます。
行6ではDataFrameに格納されて全世界のデータから日本のデータだけ絞り込んでいます。
行9ではPandasのto_datetime()関数でstr型の日付をdatetime型に変換しています。
行12では列「date」をindexに設定しています。
これで日付を指定してDataFrameを絞り込むことができます。
# Select columns
cols = ['location','date','new_cases','new_deaths','new_tests','people_vaccinated','people_fully_vaccinated','new_vaccinations']
dfx = raw[cols]
# Filter by Japan
df = dfx[ dfx['location'] == 'Japan' ].copy()
# Convert str to datetime
df['date'] = pd.to_datetime(df['date'])
# Set index
df.set_index('date', inplace=True)
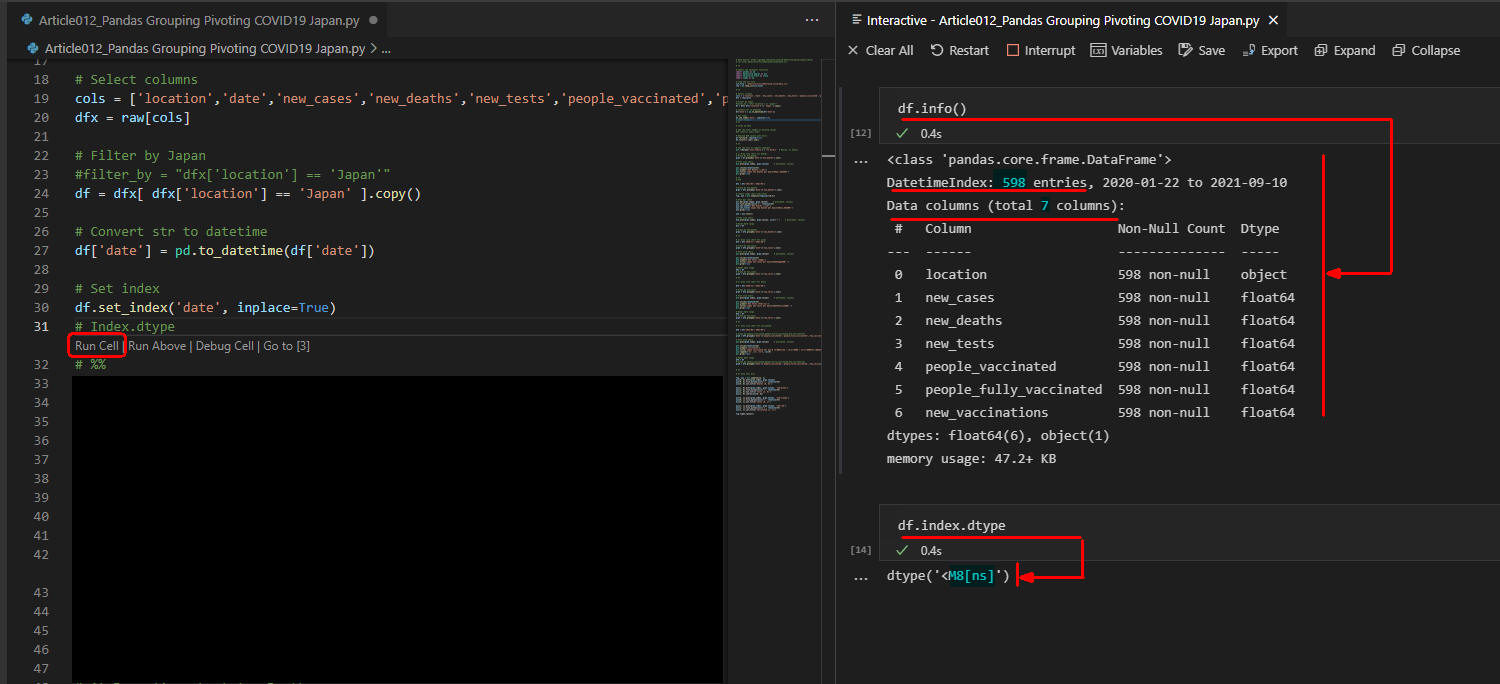 図3
右側のインタラクティブウィンドウから「df.info()」と「df.index.dtype」を入力して実行させます。
図3は実行結果です。
レコード件数が「115,872」から「598」に減少しています。
さらに列「date」のデータ型がM8[ns](datetime64と同様)に変換されています。
図3
右側のインタラクティブウィンドウから「df.info()」と「df.index.dtype」を入力して実行させます。
図3は実行結果です。
レコード件数が「115,872」から「598」に減少しています。
さらに列「date」のデータ型がM8[ns](datetime64と同様)に変換されています。
-
データをクリーンナップする
「Run Cell」をクリックしてセル内のコードを実行させます。
行7ではDataFrameのfillna()メソッドでNaN(Not a Numberの意味)の値を「0」で置換しています。
つまり、不正なデータを0にしています。
行8ではDataFrameのisnull()メソッドでNaNの件数を表示させています。
# Clean up data
# Get the total number of missing values
#df.isnull().sum().sum()
# Replace NaN values width Zeros
df.fillna(0, inplace=True)
df.isnull().sum().sum()
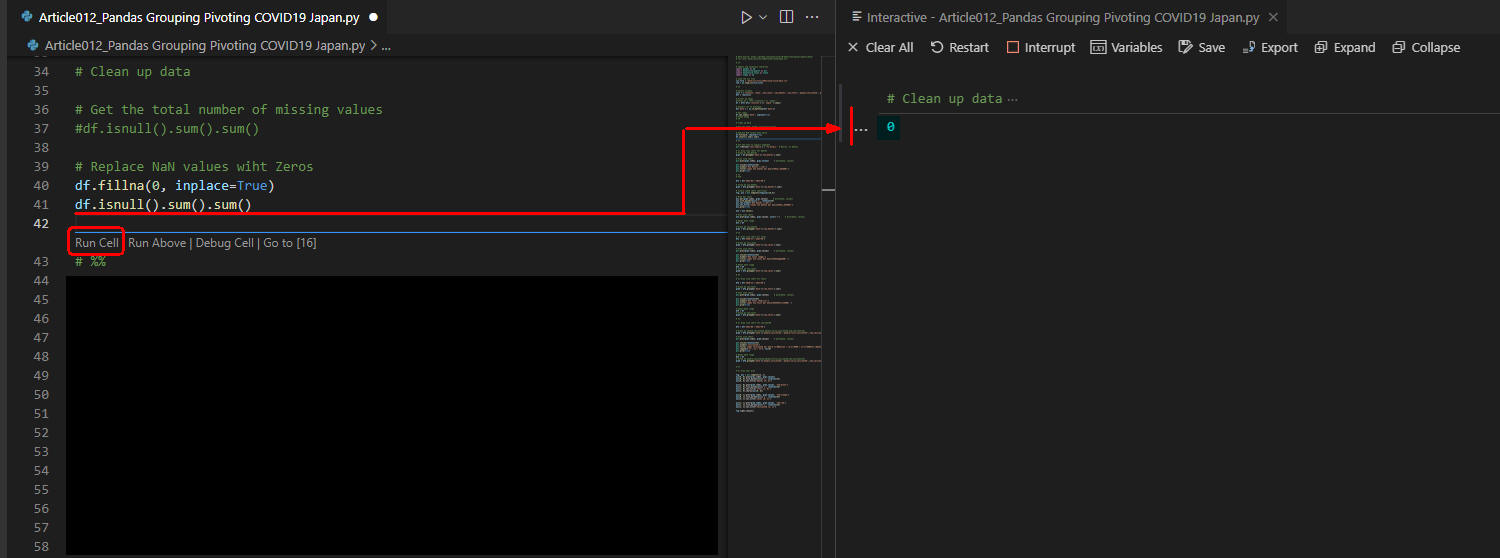 図4
図4は実行結果です。「isnull()」の件数が「0」と表示されています。
これでデータのクリーンナップが完了です。
図4
図4は実行結果です。「isnull()」の件数が「0」と表示されています。
これでデータのクリーンナップが完了です。
-
日別死亡人数のラインチャートを作成する
「Run Cell」をクリックしてセル内のコードを実行させます。
行2では日本語に対応させるために日本語のフォント「Yu Gothic」を設定しています。
行6ではDataFrameのgroupby()メソッドで列「new_deaths」を日別(date)に集計しています。
行9では図のサイズを設定しています。
行10はMatplotlibのplot()メソッドでラインチャートを作成しています。
plot()の引数1にはDataFrameのindex(date)を指定しています。
引数2にはDataFrameのvalues(new_deathsの日別集計値)を指定しています。
行12ではX軸のラベルを45度で表示させています。
行13ではY軸のラベルを設定しています。
行14ではラインチャートのタイトルを設定しています。
行15ではラインチャートにグリッドを表示させています。
# Set the font to support Japanese
plt.rcParams['font.family'] = 'Yu Gothic' # Meiryo, Yu Gothic
# 1) Draw line chart for Deaths
# Group by new_deaths
grp1 = df.groupby('date')['new_deaths'].sum()
# Plot line chart
plt.figure(figsize=(10,8))
plt.plot(grp1.index, grp1.values) #plot(date, values)
plt.xticks(rotation=45)
plt.ylabel('New Deaths (死亡)')
plt.title('Japan new deaths per day\n(日別死亡人数)')
plt.grid(True)
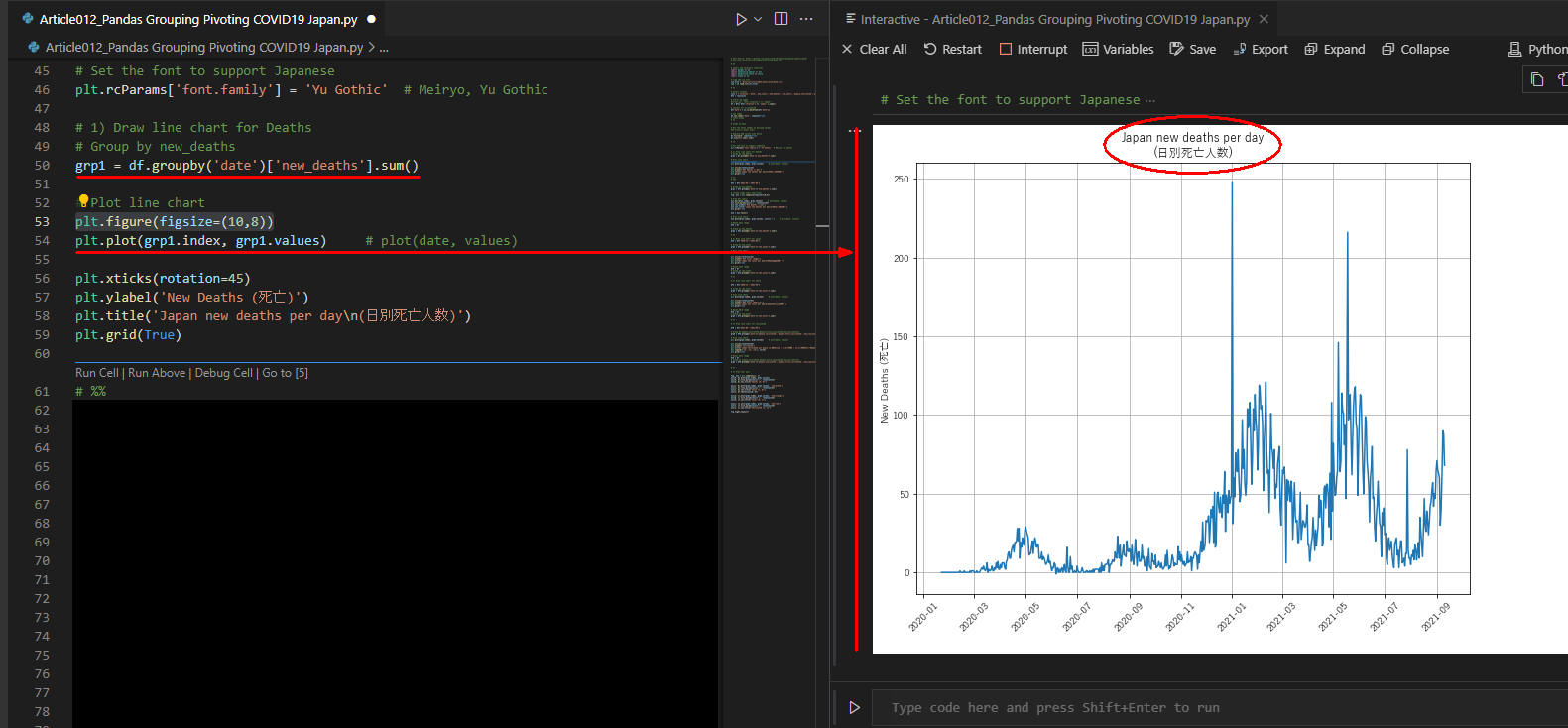 図5
図5は実行結果です。
日別の死亡人数のラインチャートが表示されています。
日付は「2020/01/01」から「2021/09/12」の範囲です。
1月と5月ごろがピークになっています。
図5
図5は実行結果です。
日別の死亡人数のラインチャートが表示されています。
日付は「2020/01/01」から「2021/09/12」の範囲です。
1月と5月ごろがピークになっています。
TIP1:バーチャートとラインチャートを重ねて描画させるには
行2ではDataFrameに絞り込み条件を設定して、チャートに表示する日付の範囲を「2021-05」~「2021-06」にしています。
行5ではDataFrameのgroupby()メソッドとsum()メソッドで日別の死亡人数を集計しています。
行8ではmatplotlibのsubplots()メソッドでfigureとsubplotのインスタンスを1個ずつ生成しています。
行11-15では日別死亡人数のバーチャートをsubplotに作成しています。
行17ではmatplotlibのaxes.twinx()メソッドで、先に作成したバーチャートとX軸を共有するチャートを作成できるsubplotを生成しています。
行20では日別の死亡人数のラインチャートをsubplotに作成しています。
バーチャートとラインチャートのsubplotはX軸を共有しているので2つのチャートが重ねて描画されます。
# Filter Date
df1 = df['2021-05':'2021-06']
# Group by new_deaths
grp1 = df1.groupby('date')['new_deaths'].sum()
# Create combo chart (bar/line)
fig, ax1 = plt.subplots(figsize=(10,6))
# Plot bar chart
ax1.bar(grp1.index, grp1.values) # plot(date, values)
ax1.tick_params(axis='x', rotation=45)
ax1.set_ylabel('New Deaths (死亡)')
ax1.set_title('Japan new deaths per day\n(日別死亡人数)')
ax1.grid(True)
ax2 = ax1.twinx()
# Plot line chart
ax2.plot(grp1.index, grp1.values, color='r') # plot(date, values)
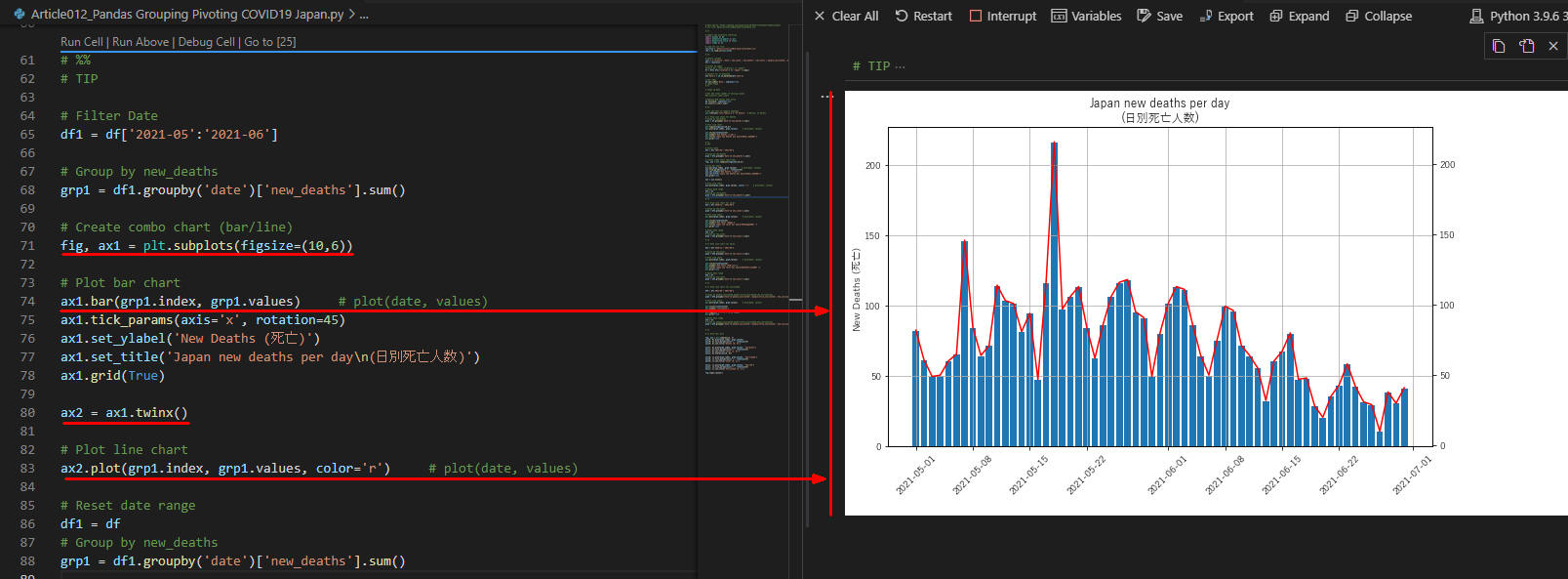 図TIP1
TIP1の実行結果です。バーチャートとラインチャートが重ねて表示されています。
図TIP1
TIP1の実行結果です。バーチャートとラインチャートが重ねて表示されています。
-
日別発症人数のラインチャートを作成する
「Run Cell」をクリックしてセル内のコードを実行させます。
行2ではDataFrameに絞り込み条件を指定してラインチャートの日付を「2020-11」~「2021-06」に絞り込んでいます。
行5ではDataFrameのgroupby()とsum()で日別発症人数を集計しています。
行9では日別発症人数のラインチャートを作成しています。
# 2) Draw line chart for cases
df2 = df['2020-11':'2021-06']
# Group by new_cases
grp2 = df2.groupby('date')['new_cases'].sum()
# Plot line chart
plt.figure(figsize=(10,4))
plt.plot(grp2.index, grp2.values) # plot(date, values)
plt.xticks(rotation=45)
plt.ylabel('New Cases (発症)')
plt.title('Japan new cases per day\n(日別発症人数)')
plt.grid(True)
# Reset date range
df2 = df
# Group by new_cases
grp2 = df2.groupby('date')['new_cases'].sum()
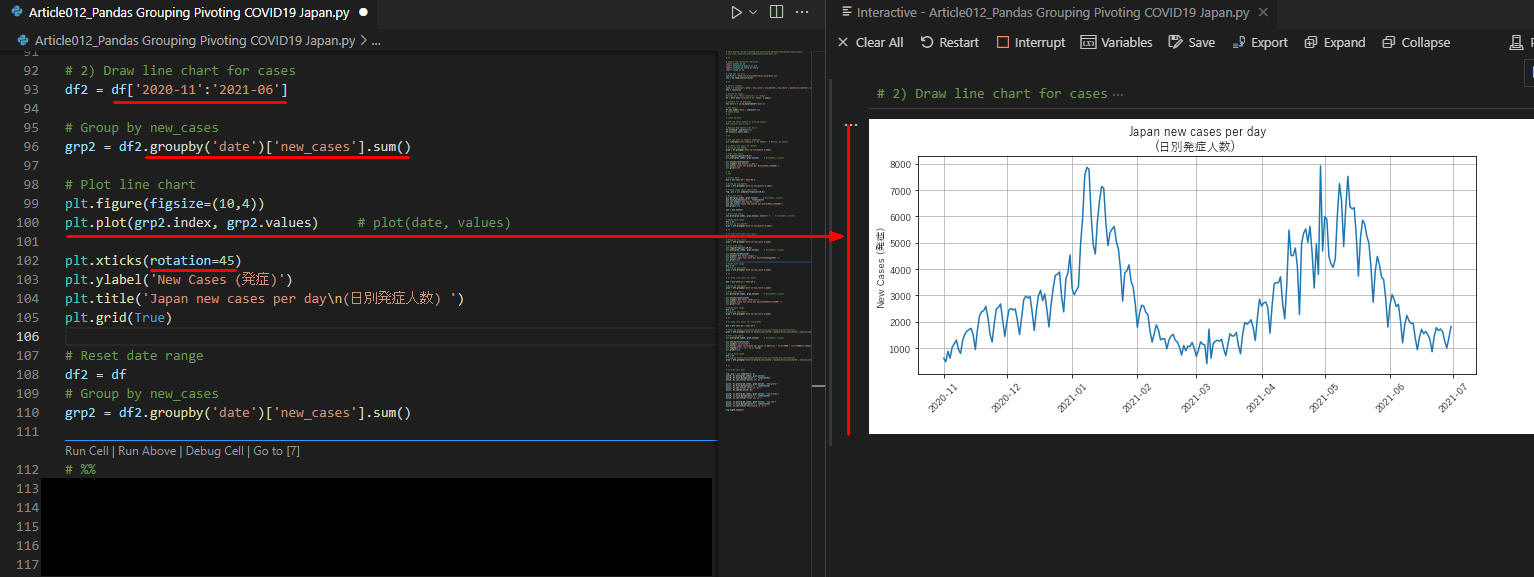 図6
図6は実行結果です。日別発症人数のラインチャートが表示されています。
2021年1月と5月ごろがピークになっているのがわかります。
図6
図6は実行結果です。日別発症人数のラインチャートが表示されています。
2021年1月と5月ごろがピークになっているのがわかります。
-
日別PCR検査人数のラインチャートを作成する
「Run Cell」をクリックしてセル内のコードを実行させます。
行2ではDataFrameに絞り込み条件を指定してラインチャートの日付を「2020-11」~「2021-09」に絞り込んでいます。
行5ではDataFrameのgroupby()とsum()で日別PCR検査人数を集計しています。
行9では日別PCR検査人数のラインチャートを作成しています。
# 3) Draw line chart for tests
df3 = df['2020-11':'2021-09']
# Group by new_tests
grp3 = df3.groupby('date')['new_tests'].sum()
# Plot line chart
plt.figure(figsize=(10,4))
plt.plot(grp3.index, grp3.values) # plot(date, values)
plt.xticks(rotation=45)
plt.ylabel('New Tests (PCR検査)')
plt.title('Japan new tests per day\n(日別PCR検査人数)')
plt.grid(True)
# Reset date range
df3 = df
# Group by new_tests
grp3 = df3.groupby('date')['new_tests'].sum()
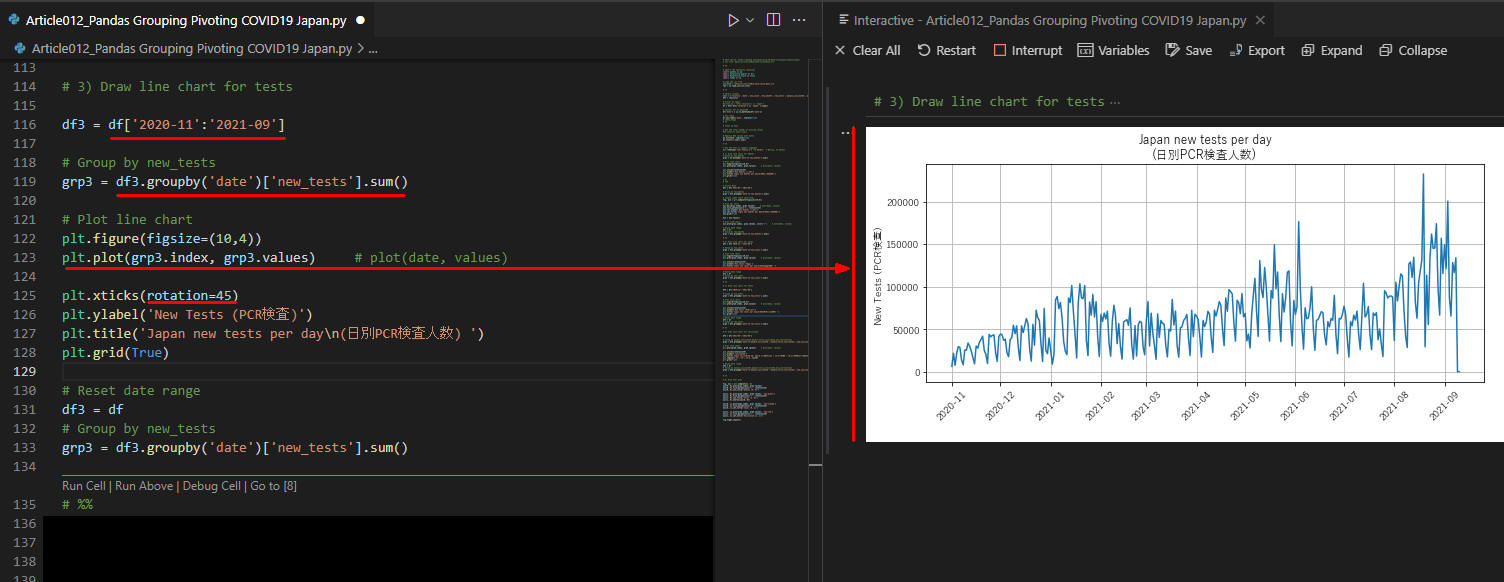 図7
図7は実行結果です。日別PCR検査人数のラインチャートが表示されています。 2021年6月と8月,9月ごろがピークになっているのがわかります。
図7
図7は実行結果です。日別PCR検査人数のラインチャートが表示されています。 2021年6月と8月,9月ごろがピークになっているのがわかります。
-
日別ワクチン接種人数のラインチャートを作成する
「Run Cell」をクリックしてセル内のコードを実行させます。
行4ではDataFrameに絞り込み条件を指定してラインチャートの日付を「2021-05」~「2021-09」に絞り込んでいます。
行9ではDataFrameのgroupby()とsum()で日別ワクチン接種人数を集計しています。
列「people_vaccinated」にはワクチン1回接種、
列「people_fully_vaccinated」にはワクチン2回接種の人数が格納されています。
groupby()で複数の列を集計するとき、行8のように記述すると警告が表示されます。
警告を無視したいときは行2の「import warnings」と、行7の「warnings.simplefilter('ignore')」を追加します。
元の状態に戻したいときは行10の「warnings.resetwarnings()」を追加します。
ちなみに警告が出ないようにするには行9のように記述します。
行14では日別ワクチン接種人数のラインチャートを作成しています。
# 4) Draw line chart for vaccinated
import warnings
df4 = df['2021-05':'2021-09']
# Group by people_vaccinated,people_fully_vaccinated,new_vaccinations
#warnings.simplefilter('ignore')
#grp4 = df4.groupby('date')['people_vaccinated','people_fully_vaccinated'].sum()
grp4 = df4.groupby('date')[['people_vaccinated','people_fully_vaccinated']].apply(sum)
#warnings.resetwarnings()
# Plot line chart
plt.figure(figsize=(10,4))
plt.plot(grp4.index, grp4.values)
plt.xticks(rotation=45)
plt.ylabel('vaccinated')
plt.title('Japan vaccinated per day\n v1:1回接種 / v2:2回接種\n(ワクチン接種状況)')
plt.legend(['v1','v2'], loc=0)
plt.grid(True)
# Reset date range
df4 = df
grp4 = df4.groupby('date')[['people_vaccinated','people_fully_vaccinated']].apply(sum)
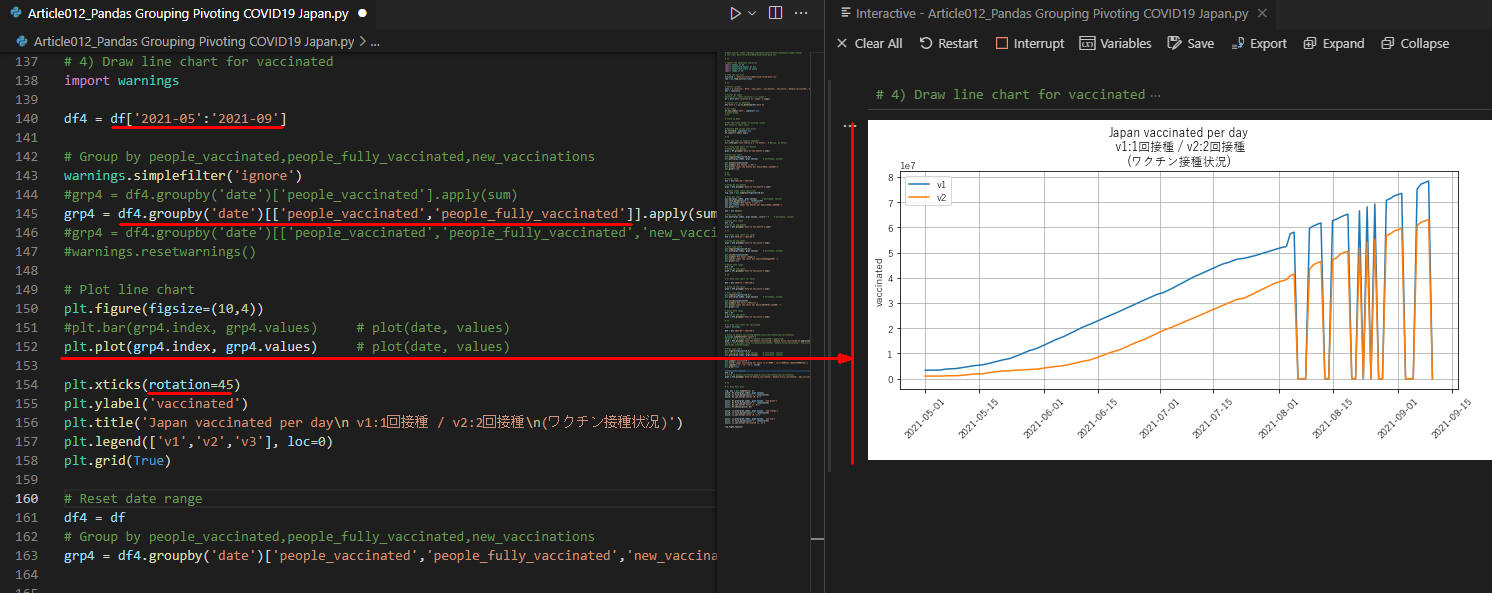 図8
図8は実行結果です。2021年の5月ごろから右肩上がりでワクチンの接種が増えていることが分かります。
図8
図8は実行結果です。2021年の5月ごろから右肩上がりでワクチンの接種が増えていることが分かります。
-
4種類のラインチャートをまとめて描画する
「Run Cell」をクリックしてセル内のコードを実行させます。
行3ではmatplotlibのsubplots()メソッドで1個のfigureと4個のsubplotのインスタンスを生成しています。
行5-7ではsubplotに日別死亡人数のラインチャートを上段の左側に描画されるように作成しています。
行9-12ではsubplotに日別PCR検査人数のラインチャートを下段の左側に描画されるように作成しています。
行14-16ではsubplotに日別発症人数のラインチャートを上段の右側に描画されるように作成しています。
行18-20ではsubplotに日別接種人数のラインチャートを下段の右側に描画されるように作成しています。
行22ではfigureのtight_layout()メソッドでそれぞれのsubplotの間隔が自動的に調整されるようにしています。
# 5) Draw Four plots
fig, axs = plt.subplots(2, 2, figsize=(10,6))
axs[0, 0].plot(grp1.index, grp1.values)
axs[0, 0].tick_params(axis='x', rotation=45)
axs[0, 0].set_title('Deaths(死亡) [0, 0]')
axs[1, 0].plot(grp3.index, grp3.values, 'tab:green')
axs[1, 0].tick_params(axis='x', rotation=45)
axs[1, 0].set_title('Tests(PCR検査) [1, 0]')
axs[1, 0].sharex(axs[0, 0])
axs[0, 1].plot(grp2.index, grp2.values, 'tab:orange')
axs[0, 1].tick_params(axis='x', rotation=45)
axs[0, 1].set_title('Cases(発症) [0, 1]')
axs[1, 1].plot(grp4.index, grp4.values, 'tab:red')
axs[1, 1].tick_params(axis='x', rotation=45)
axs[1, 1].set_title('Vaccinated(接種) [1, 1]')
fig.tight_layout()
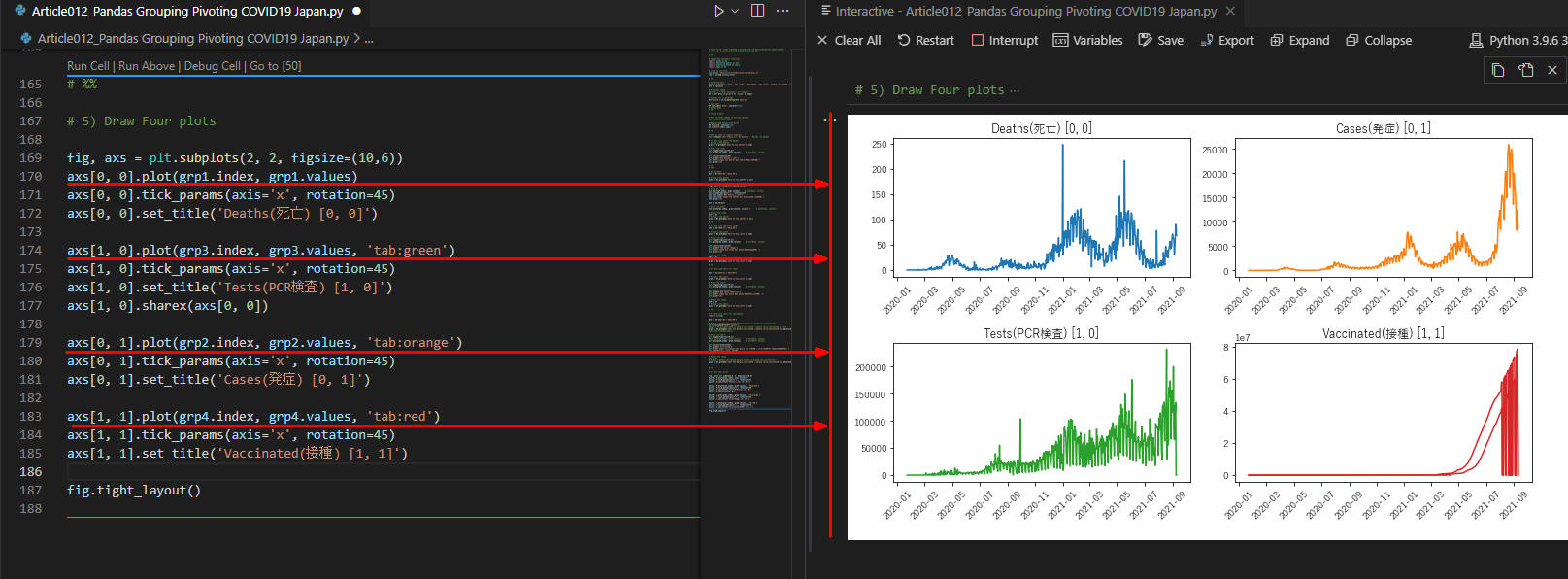 図9
図9は実行結果です。
figure(図)に4種類のラインチャートが表示されています。
図9
図9は実行結果です。
figure(図)に4種類のラインチャートが表示されています。
-
すべてのコードをまとめて掲載
最後にこれまで解説したすべてのコードをまとめて掲載しましたので参考にしてください。
# Data Source: https://github.com/owid/covid-19-data/tree/master/public/data/
# csv file: data/csv/article012/owid-covid-data.csv
# %%
# Import the necessary libraries
import pandas as pd
import matplotlib.pyplot as plt
import matplotlib.style as style
import numpy as np
# Load the csv file
csv_file = 'data/csv/article012/owid-covid-data.csv'
raw = pd.read_csv(csv_file)
# %%
# Select columns
cols = ['location','date','new_cases','new_deaths','new_tests','people_vaccinated','people_fully_vaccinated','new_vaccinations']
dfx = raw[cols]
# Filter by Japan
#filter_by = "dfx['location'] == 'Japan'"
df = dfx[ dfx['location'] == 'Japan' ].copy()
# Convert str to datetime
df['date'] = pd.to_datetime(df['date'])
# Set index
df.set_index('date', inplace=True)
# Index.dtype
# %%
# Clean up data
# Get the total number of missing values
#df.isnull().sum().sum()
# Replace NaN values wiht Zeros
df.fillna(0, inplace=True)
df.isnull().sum().sum()
# %%
# Set the font to support Japanese
plt.rcParams['font.family'] = 'Yu Gothic' # Meiryo, Yu Gothic
# 1) Draw line chart for Deaths
# Group by new_deaths
grp1 = df.groupby('date')['new_deaths'].sum()
# Plot line chart
plt.figure(figsize=(10,8))
plt.plot(grp1.index, grp1.values) # plot(date, values)
plt.xticks(rotation=45)
plt.ylabel('New Deaths (死亡)')
plt.title('Japan new deaths per day\n(日別死亡人数)')
plt.grid(True)
# %%
# TIP1
# Filter Date
df1 = df['2021-05':'2021-06']
# Group by new_deaths
grp1 = df1.groupby('date')['new_deaths'].sum()
# Create combo chart (bar/line)
fig, ax1 = plt.subplots(figsize=(10,6))
# Plot bar chart
ax1.bar(grp1.index, grp1.values) # plot(date, values)
ax1.tick_params(axis='x', rotation=45)
ax1.set_ylabel('New Deaths (死亡)')
ax1.set_title('Japan new deaths per day\n(日別死亡人数)')
ax1.grid(True)
ax2 = ax1.twinx()
# Plot line chart
ax2.plot(grp1.index, grp1.values, color='r') # plot(date, values)
# Reset date range
df1 = df
# Group by new_deaths
grp1 = df1.groupby('date')['new_deaths'].sum()
# %%
# 2) Draw line chart for cases
df2 = df['2020-11':'2021-06']
# Group by new_cases
grp2 = df2.groupby('date')['new_cases'].sum()
# Plot line chart
plt.figure(figsize=(10,4))
plt.plot(grp2.index, grp2.values) # plot(date, values)
plt.xticks(rotation=45)
plt.ylabel('New Cases (発症)')
plt.title('Japan new cases per day\n(日別発症人数) ')
plt.grid(True)
# Reset date range
df2 = df
# Group by new_cases
grp2 = df2.groupby('date')['new_cases'].sum()
# %%
# 3) Draw line chart for tests
df3 = df['2020-11':'2021-09']
# Group by new_tests
grp3 = df3.groupby('date')['new_tests'].sum()
# Plot line chart
plt.figure(figsize=(10,4))
plt.plot(grp3.index, grp3.values) # plot(date, values)
plt.xticks(rotation=45)
plt.ylabel('New Tests (PCR検査)')
plt.title('Japan new tests per day\n(日別PCR検査人数) ')
plt.grid(True)
# Reset date range
df3 = df
# Group by new_tests
grp3 = df3.groupby('date')['new_tests'].sum()
# %%
# 4) Draw line chart for vaccinated
import warnings
df4 = df['2021-05':'2021-09']
# Group by people_vaccinated,people_fully_vaccinated,new_vaccinations
warnings.simplefilter('ignore')
#grp4 = df4.groupby('date')['people_vaccinated','people_fully_vaccinated'].sum()
grp4 = df4.groupby('date')[['people_vaccinated','people_fully_vaccinated']].apply(sum)
#warnings.resetwarnings()
# Plot line chart
plt.figure(figsize=(10,4))
#plt.bar(grp4.index, grp4.values) # plot(date, values)
plt.plot(grp4.index, grp4.values) # plot(date, values)
plt.xticks(rotation=45)
plt.ylabel('vaccinated')
plt.title('Japan vaccinated per day\n v1:1回接種 / v2:2回接種\n(ワクチン接種状況)')
plt.legend(['v1','v2'], loc=0)
plt.grid(True)
# Reset date range
df4 = df
# Group by people_vaccinated,people_fully_vaccinated,new_vaccinations
grp4 = df4.groupby('date')[['people_vaccinated','people_fully_vaccinated']].apply(sum)
# %%
# 5) Draw Four plots
fig, axs = plt.subplots(2, 2, figsize=(10,6))
axs[0, 0].plot(grp1.index, grp1.values)
axs[0, 0].tick_params(axis='x', rotation=45)
axs[0, 0].set_title('Deaths(死亡) [0, 0]')
axs[1, 0].plot(grp3.index, grp3.values, 'tab:green')
axs[1, 0].tick_params(axis='x', rotation=45)
axs[1, 0].set_title('Tests(PCR検査) [1, 0]')
axs[1, 0].sharex(axs[0, 0])
axs[0, 1].plot(grp2.index, grp2.values, 'tab:orange')
axs[0, 1].tick_params(axis='x', rotation=45)
axs[0, 1].set_title('Cases(発症) [0, 1]')
axs[1, 1].plot(grp4.index, grp4.values, 'tab:red')
axs[1, 1].tick_params(axis='x', rotation=45)
axs[1, 1].set_title('Vaccinated(接種) [1, 1]')
fig.tight_layout()